家蚕基因组数据库更新版(SilkDB3.0)上线
王翊教授课题组发布家蚕多组学分析和可视化在线平台
近日,西南大学蚕丝科学与技术研究团队的王翊教授课题组在生物领域国际顶级期刊《Nucleic Acids Research》(IF: 11.561)在线发表了题为“SilkDB3.0: visualizing and exploring multiple levels of data for silkworm”的原创性研究论文,公布了家蚕基因组数据跟新版SilkDB3.0,并同时发布了一个家蚕的多组学数据分析并生成可视化结果的在线平台SilkDB3.0(https://silkdb.bioinfotoolkits.net)。这是家蚕功能基因组大数据分析团队在开发OrthoVenn2后,半年内发表的第二篇高水平生物信息学文章。
家蚕(Bombyx mori)是一种重要的经济昆虫,也是鳞翅目的代表,具有昆虫学研究和遗传学分析的重要价值,整合家蚕多组学数据并进化可视化展示对家蚕研究具有重要意义,但是还没有平台可以提供家蚕多个水平的数据,并提供搜索,分析和可视化功能。此前,团队分别于2005年发表了SilkDB,其更新版本(SilkDB 2.0)于2010年发布,两次论文均发表在《Nucleic Acids Research》期刊。近十年来,关于家蚕的研究产生了的海量数据,包括基因组,转录组,Hi-C(高通量染色质构象捕获)和pangenome(泛基因组)。为了更好地整合这些数据并开发用户友好的可视化界面,团队将SilkDB更新到了3.0版本,该版本与以前的版本相比具有实质性的性能改进,包括搜索多组学数据,可视化界面和更多的工具,王翊教授领衔师生具体完成了SilkDB3.0的设计与维护。
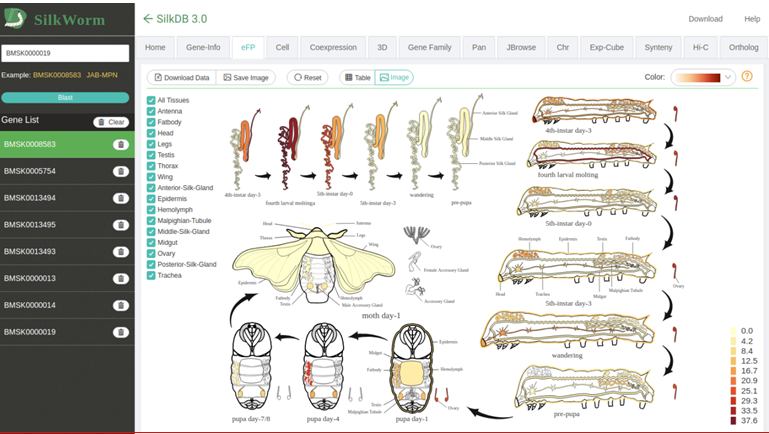
在SilkDB3.0中,基因组的组装质量得到显著提高,包含重新注释的编码基因以及大量转录组数据,可用于探索家蚕基因在不同组织中的表达。该数据平台还包含了来自不同地理位置的163个家蚕和野蚕样品的泛基因组数据,从而能更全面地描述家蚕的遗传变异(单核苷酸多态性和插入缺失)。同时,平台还提供了家蚕六种不同组织的Hi-C数据,以帮助理解基因调控机制。SilkDB3.0主要分为以下模块:基因信息,表达谱数据可视化,亚细胞定位预测,共表达网络,蛋白质3D结构,基因家族,pangenome,JBrowse,染色体定位,共线性分析,Hi-C和同源性分析。SilkDB3.0不仅包含基因组序列分析的基本功能,还实现了多个数据可视化工具的有机整合,通过用户友好的交互式界面全面展示了家蚕多组学数据,对家蚕和其他昆虫研究具有重要意义。SilkDB3.0版本的发布,标志着我校家蚕基因组学研究全面进入多组学时代,对未来的家蚕研究具有重要的基础性支撑意义。自数据库上线以来,受到国际国内同行的广泛关注。
西南大学为该论文成果的唯一完成单位,大数据工程师鲁方和硕士二年级学生魏昭渊为论文的第一作者,王翊教授为论文的通讯作者。SilkDB1.0、2.0版第一和通讯作者夏庆友教授为本文的作者之一,参与了3.0版本的研发,并在数据库顶层设计和运算要求上发挥了重要作用,使得数据保持了既往优势。文章同时致谢生物大数据分析团队的成员和为该研究作出贡献的所有参与者。该项研究得到了国家自然科学基金委面上项目、中央高校团队培育项目、西南大学生物学“双一流”建设项目以及“聚贤工程”人才启动项目等基金资助。王翊教授还受到国家海外高层次人才青年项目以及重庆市留学生创新创业计划项目支持。
原文链接:doi.org/10.1093/nar/gkz919
供 稿:陈玉琳